研究分野の概略
生物のデザインを決めるゲノムDNAは、地球上で最も重要な情報であるにも関わらず壊れやすい物質です。当研究室では、ゲノムの再生メカニズムとその不調が引き起こす細胞老化、がん化の機構を研究しています。
研究内容の紹介
ゲノムの再生メカニズムとその不調が引き起こす細胞老化、がん化機構
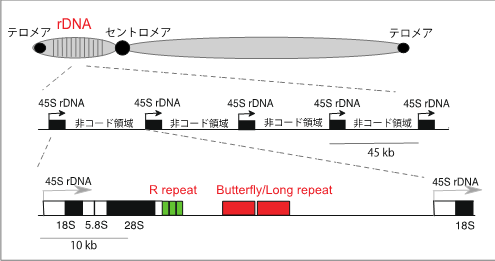
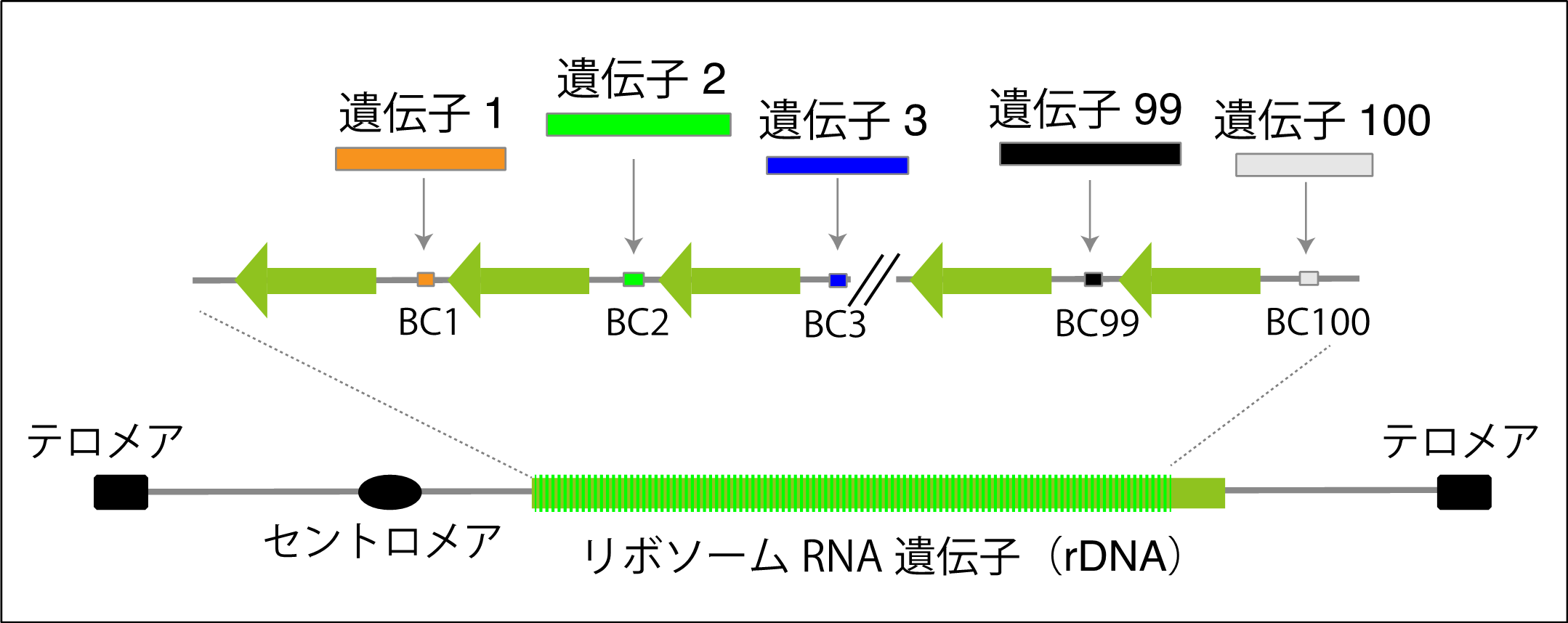
遺伝情報を担うゲノムDNAは化学物質や放射線に対して極めて弱い物質てす。生物は 進化の過程で、この弱いDNAを再生し、生命の連続性を維持する能力を獲得してきました。 しかし完全に直すことは不可能で、少しすつ「変異」が蓄積し、細胞は徐々に老化していきます。変異が細胞増殖に関わる遺伝子に生じた場合にはがん化することもあります。がん化はその細胞だけの問題ではなく、個体が死に追いやられることもあります。寿命が長い生殖細胞や幹細胞ではゲノムの維持が特に重要です。生殖細胞は次世代へ命をつなぐ重要な細胞です。また幹細胞も個体が生きている限り分裂し、新しい細胞を供給し続けるため、変異の蓄積は極力避ける必要があります。これらの細胞ではゲノムの「修復」では追いつかず、「再生」レベルの維持・管理が必要となります。我々の研究室では酵母、マウス、ヒト細胞を用いて、ゲノム再生の分子機構を特にゲノム中で安定性が低い反復配列「リボソームRNA遺伝子(rDNA)」に注目し研究しています。 さらにゲノムの不安定化を軽減し、老化およびがん化抑制に働く物質の探索も行なっています。共同研究等も幅広く行なっています。
論文一覧
- Ribosomal RNA gene repeats, their stability and cellular senescence.
Kobayashi T.
Proc Jpn Acad Ser B Phys Biol Sci. 2014 Apr 11;90(4): 119-129. doi: 10.2183/pjab.90.119.
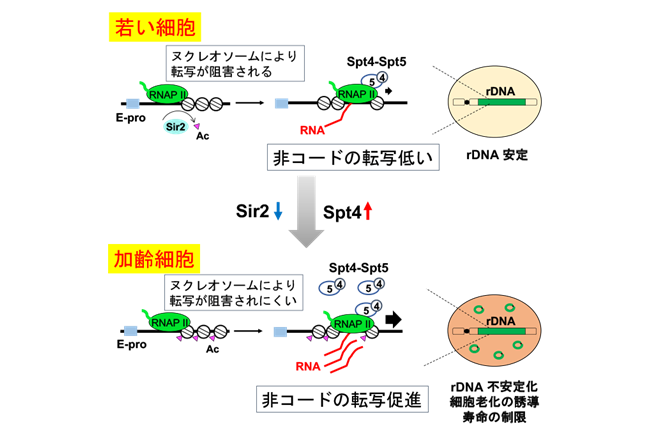
- Cellular Senescence in Yeast Is Regulated by rDNA Noncoding Transcription.
Saka K, Ide S, Ganley AR, Kobayashi T.
Curr Biol. 2013 Sep 23;23(18):1794-8. doi: 10.1016/j.cub.2013.07.048.
- Regulation of ribosomal RNA gene copy number and its role in modulating genome integrity and evolutionary adaptability in yeast.
Kobayashi T.
Cell Mol Life Sci. 2011 Apr;68(8):1395-403. doi: 10.1007/s00018-010-0613-2.
- Abundance of ribosomal RNA gene copies maintains genome integrity.
Ide S, Miyazaki T, Maki H, Kobayashi T.
Science. 2010 Feb 5;327(5966):693-6. doi: 10.1126/science.1179044.
- The effect of replication initiation on gene amplification in the rDNA and its relationship to aging.
Ganley ARD, Ide S, Saka K, Kobayashi T.
Mol Cell. 2009 Sep 11;35(5):683-93. doi: 10.1016/j.molcel.2009.07.012.
- Recombination regulation by transcription-induced cohesin dissociation in rDNA repeats.
Kobayashi T and Ganley ARD.
Science. 2005 Sep 2;309(5740):1581-4. doi: 10.1126/science.1116102.
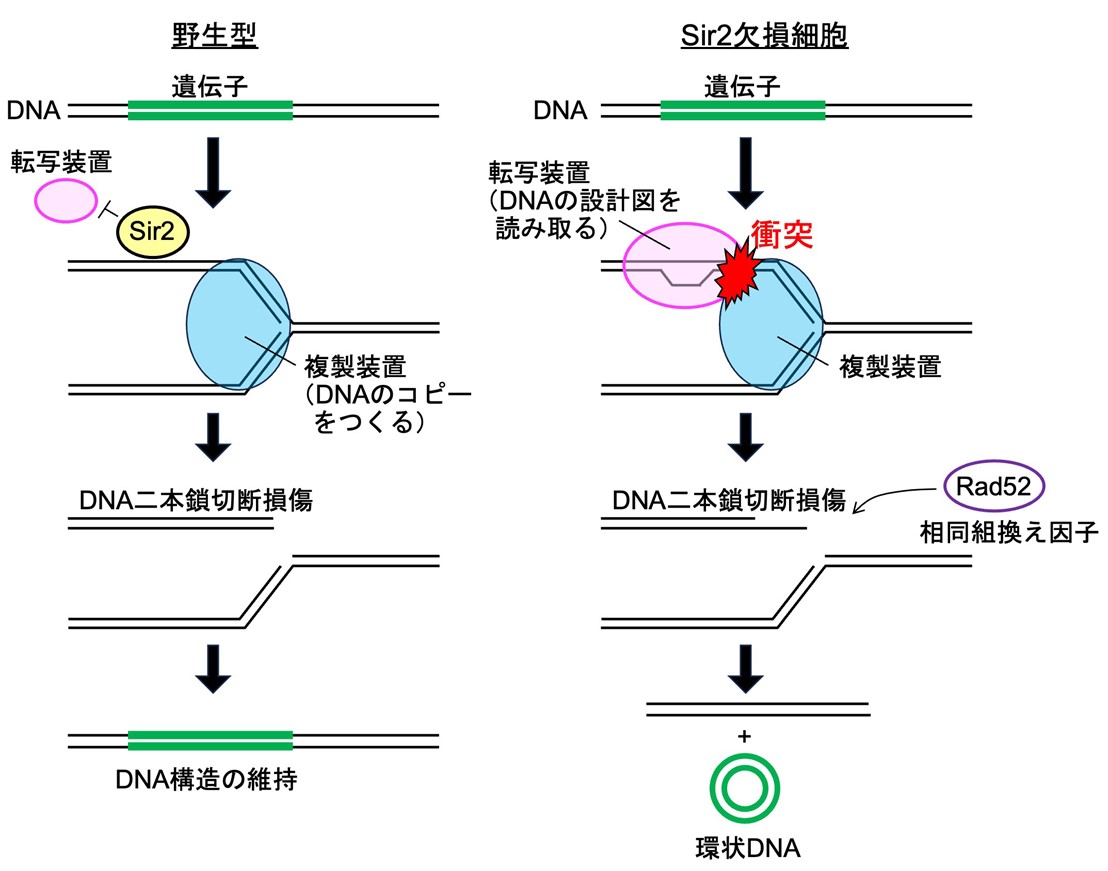
- SIR2 regulates recombination between different rDNA repeats, but not recombination within individual rRNA genes in yeast.
Kobayashi T, Horiuchi T, Tongaonkar P, Vu L, Nomura M .
Cell. 2004 May 14;117(4):441-53. doi: 10.1016/S0092-8674(04)00414-3
- Transcription-dependent recombination and the role of fork collision in yeast rDNA.
Takeuchi Y, Horiuchi T, Kobayashi T.
Genes Dev. 2003 Jun 15;17(12):1497-506. doi: 10.1101/gad.1085403
- Expansion and contraction of ribosomal DNA repeats in Saccharomyces cerevisiae: requirement of replication fork blocking (Fob1) protein and the role of RNA polymerase I.
Kobayashi T, Heck DJ, Nomura M, Horiuchi T.
Genes Dev. 1998 Dec 15;12(24):3821-30. doi:10.1101/gad.12.24.3821
- Evidence of a ter specific binding protein essential for the termination reaction of DNA replication in Escherichia coli.
Kobayashi T, Hidaka M, Horiuchi T.
EMBO J. 1989 Aug;8(8):2435-41..
小林 武彦 教授
KOBAYASHI Takehiko
博士(理学)
理学系研究科・生物科学専攻
大屋 恵梨子 助教
OYA Eriko
博士(理学)
堀 優太郎 助教
HORI Yutaro
博士(理学)
柳 秀一 特任助教
YANAGI Shuichi
博士(理学)
朝倉 智子 技術専門職員
ASAKURA Tomoko