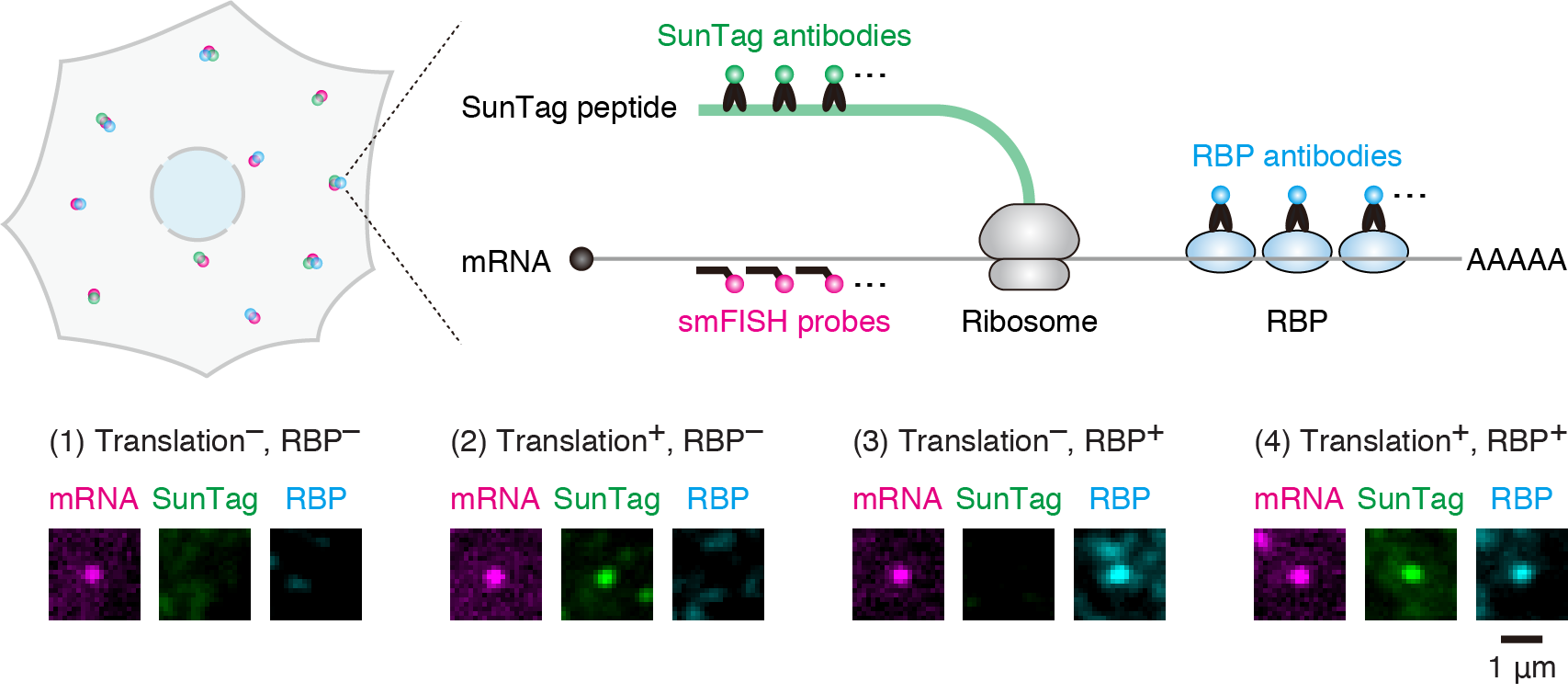
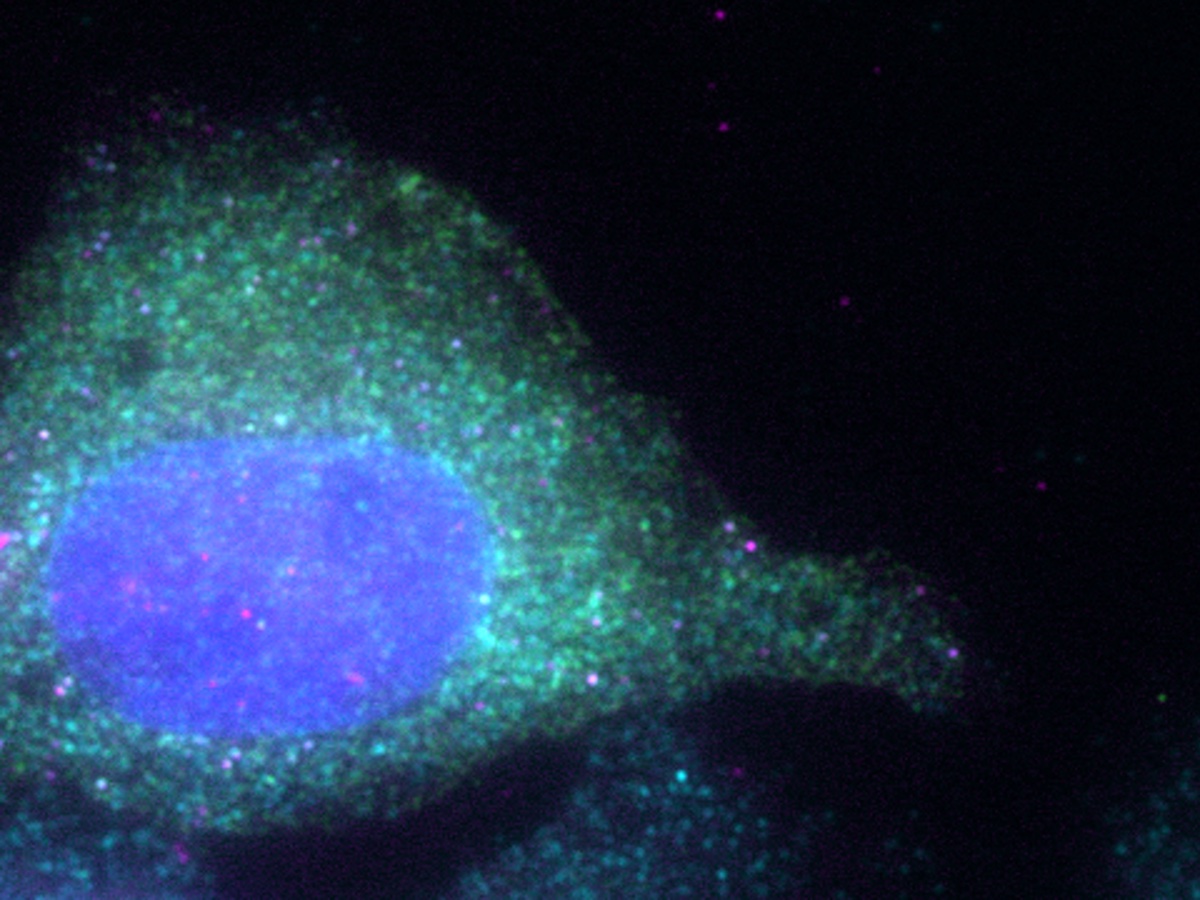
研究分野の概略
私たちの研究分野では、mRNAから蛋白質が作られる過程、「翻訳」について研究を行っています。翻訳を1細胞・1分子mRNAの解像度で「見る」ことで、その隠された秘密を「知り」、ひいては自在に「操る」ことを目指しています。
研究内容の紹介
mRNAの翻訳を、1細胞・1分子レベルで「見る」「知る」「操る」
ヒトのゲノムDNAには、おおよそ20,000種類の遺伝子(正確には蛋白質コード遺伝子)が書き込まれています。これらの遺伝子は、DNAの配列情報がmRNAへと写し取られ(転写)、そのmRNAの配列情報をもとに蛋白質が作られることで(翻訳)、それぞれの機能を発現します。私たちの研究分野では、この遺伝子発現の後半部分、「翻訳」について研究を行っています。
翻訳は生命の基盤となる現象であり、これまで半世紀以上にわたり世界中で研究が行われてきました。ですが、実はその多くは生化学的な実験手法(例えば、細胞をすり潰し、回収したmRNAを試験管内で解析するような手法)によるものです。そのため、翻訳がどういった因子によってどのように制御されているのかという「Who・How」については理解が進んでいる一方、それが細胞内において一体いつ・どこで起こっているのかという「When・Where」についてはいまだ謎に包まれています。さらに、これまでの生化学的な実験手法では、数多くの細胞から回収した数多くのmRNAがまとめて解析され、翻訳の「総量」が測定されてきました。そのため、細胞一つ一つ、mRNA一つ一つがどういった翻訳の挙動を示すのかという点も長年にわたり見落とされています。
そこで私たちは、こうした翻訳の謎に迫るべく、翻訳の制御過程を(試験管内ではなく)細胞内において、(数多くの細胞・数多くのmRNAの総和ではなく)1細胞・1分子mRNAの解像度で可視化できる新しいイメージング技術を作りました。私たちの研究分野では、この細胞内1分子イメージング技術を自分たちの武器として、これまでにない解像度で翻訳を実際に「見る」ことで、その隠された秘密(翻訳の時空間的な側面、1細胞・1分子mRNAの挙動など)を「知る」べく研究を行っています。また、こうした基礎研究から得られる知見を活かし、新型コロナウイルスのパンデミックで脚光を浴びたmRNAワクチンの改良につなげるなど、ひいては翻訳を自在に「操る」ことを目指しています。
論文一覧
- Single-molecule imaging of microRNA-mediated gene silencing in cells.
Hotaka Kobayashi (責任著者), Robert H Singer.
Nature Communications. 13(1):1435. 2022
- Identification of an AGO (Argonaute) protein as a prey of TER94/VCP.
Hotaka Kobayashi (責任著者), Yukihide Tomari.
Autophagy. 16(1):190-192. 2020
- VCP machinery mediates autophagic degradation of empty Argonaute.
Hotaka Kobayashi (責任著者), Keisuke Shoji, Kaori Kiyokawa, Lumi Negishi, Yukihide Tomari.
Cell Reports. 28(5):1144-1153. 2019
- Iruka eliminates dysfunctional Argonaute by selective ubiquitination of its empty state.
Hotaka Kobayashi (責任著者), Keisuke Shoji, Kaori Kiyokawa, Lumi Negishi, Yukihide Tomari.
Molecular Cell. 73(1):119-129. 2019
- RISC assembly: Coordination between small RNAs and Argonaute proteins.
Hotaka Kobayashi, Yukihide Tomari.
Biochimica et Biophysica Acta. 1859(1):71-81. 2016
- Measurement of Rab35 activity with the GTP-Rab35 trapper RBD35.
Hotaka Kobayashi, Kan Etoh, Soujiro Marubashi, Norihiko Ohbayashi, Mitsunori Fukuda.
Methods in Molecular Biology. 1298:207-216. 2015
- Rab35 promotes the recruitment of Rab8, Rab13, and Rab36 to recycling endosomes through MICAL-L1 during neurite outgrowth.
Hotaka Kobayashi, Kan Etoh, Norihiko Ohbayashi, Mitsunori Fukuda.
Biology Open. 3(9):803-814. 2014
- Arf6, Rab11, and transferrin receptor define distinct populations of recycling endosomes.
Hotaka Kobayashi, Mitsunori Fukuda.
Communicative & Integrative Biology. 5:e25036. 2013
- Rab35 establishes the EHD1-association site by coordinating two distinct effectors during neurite outgrowth.
Hotaka Kobayashi, Mitsunori Fukuda.
Journal of Cell Science. 126(11):2424-2435. 2013
- Rab35 regulates Arf6 activity through centaurin-β2 (ACAP2) during neurite outgrowth.
Hotaka Kobayashi, Mitsunori Fukuda.
Journal of Cell Science. 125(9):2235-2243. 2012
小林 穂高 客員准教授
KOBAYASHI Hotaka
博士(生命科学)