研究分野の概略
私たちはDNAやRNAの自己組織化をデザインし、生体内の構造モチーフや酵素反応を活かしてナノスケールの機械やセンサーなど高度な分子システムを構築します。合成生物学や生物物理学のための有用なバイオツールの創出を通じて、生命現象の解明を目指します。
研究内容の紹介
DNAとRNAを自在に操り、新たな分子ツールで次世代バイオを切り拓く
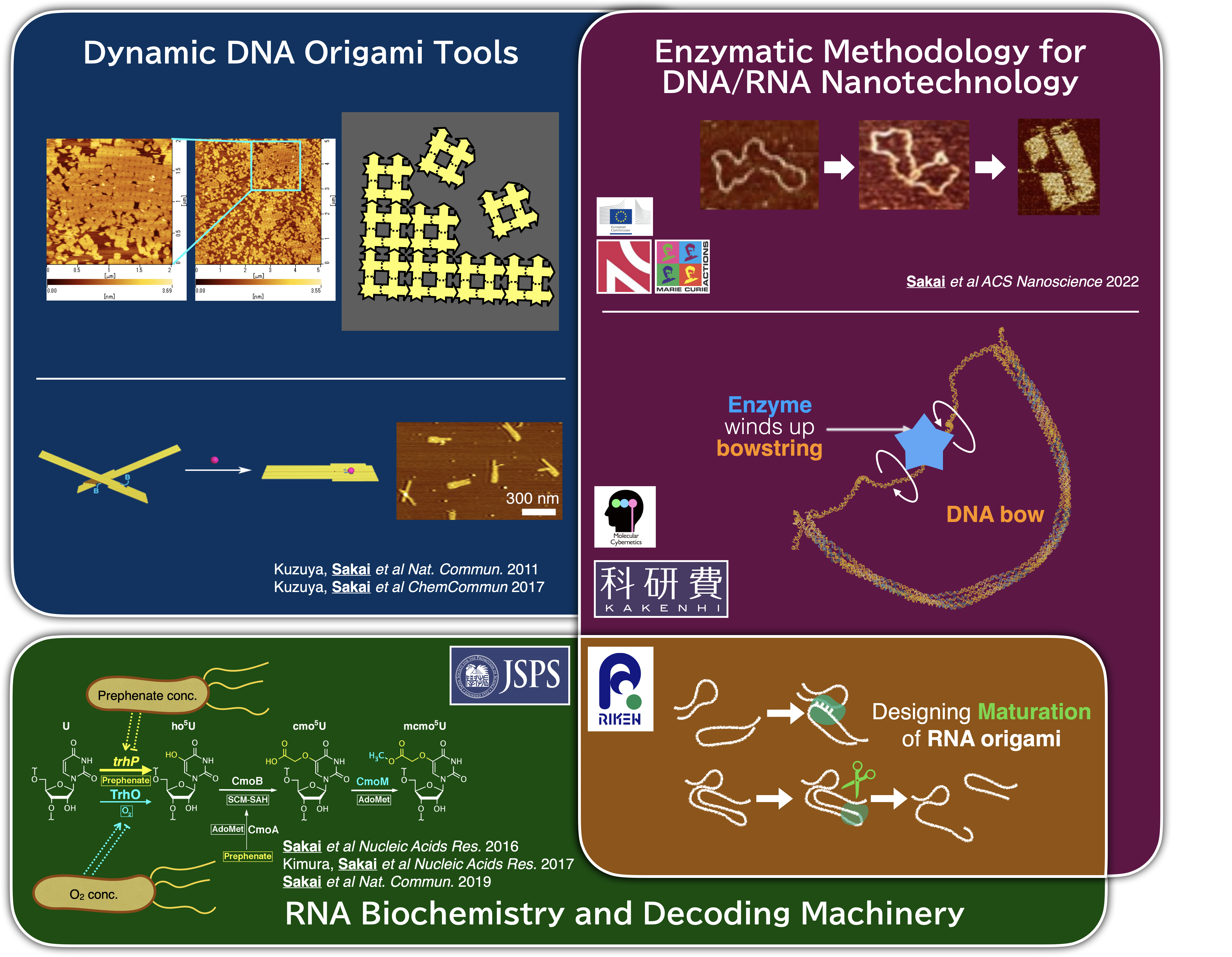
DNAやRNAといった核酸は単に遺伝情報の保管や実行を担っているだけでなく、分子レベルでの正確な自己組織化や相互作用を自在に設計できる優れた材料です。私たちは、この特性を活かした「核酸ナノテクノロジー」を研究の軸とし、DNAやRNAの自己集合をデザインしてナノスケールの機械やセンサーなど高度な分子ロボットを構築しています。
これまで、動的に構造が変化するDNAデバイスを用いたモジュール式センサーや、内部を外界から遮蔽するナノカプセル、「大面積」にわたって分子配列を実装するためのDNAオリガミ基盤などを開発してきました。特に、生命が進化の過程で洗練させてきた酵素の力に注目し、核酸構造体との統合的な活用に興味を持っています。これまでに、DNAトポイソメレースを活用することで長鎖DNAをカテナン化し、トポロジー的に連結されたDNAオリガミ構造を組み立てる方法を確立しました。現在は、別のDNAトポイソメレースをナノデバイスの駆動力として応用する研究や、細胞内に豊富なリボソーム生合成因子を利用して高度なRNAオリガミを生体内で構築する試みにも取り組んでいます。
私たちは合成生物学や生物物理学をはじめ、幅広い領域との連携し、現在、理化学研究所、東京科学大学、東京農工大学、ダラム大学(英国)などと共同研究を進めています。また、大量のオリゴDNAやそれらの混合パターンを管理するデータベースソフトウェアや実験自動化装置を導入し、それらの共同研究や、さらなる大規模な分子システムを効率的に扱うための基盤整備にも注力しています。最終的には、汎用性と拡張性を兼ね備えた分子ツールを創出し、生命現象の深層に迫ることを目指しています。なお、本研究分野は、東京大学とカロリンスカ研究所の共同プログラム「UTokyo-KI LINK」の枠組みの下、カロリンスカ研究所のビョルン・ヘーグベリ教授の研究室を拠点として研究を推進しています。
論文一覧
- Gerrit D. Wilkens, Piotr Stepien, Yusuke Sakai, Sirajul Md. Islam, Jonathan G Heddle*. “A DNA origami bubble blower for liposome production.” ACS Omega, 9, 43, 43609-43615 (2024) DOI: 10.1021/acsomega.4c05297
- Yusuke Sakai,† Gerrit D. Wilkens,† Karol Wolski, Szczepan Zapotoczny, Jonathan G Heddle*. “TOPOGAMI: Topologically linked DNA origami.” ACS Nanoscience Au, 2, 1, 57-63 (2022) DOI: 10.1021/acsnanoscienceau.1c00027 († equal contribution)
- Yusuke Sakai,† Satoshi Kimura,† Tsutomu Suzuki. “Dual pathways of tRNA hydroxylation ensure efficient translation by expanding decoding capability.” Nature Communications, 10, 2858 (2019) DOI: 10.1038/s41467-019-10750-8 († equal contribution)
- Yusuke Sakai, Md. Sirajul Islam, Martyna Adamiak, Simon Chi-Chin Shiu, Julian Alexander Tanner, Jonathan Gardiner Heddle,* “DNA Aptamers for Functionalisation of DNA Origami Nanostructures.” Genes, 9, 571 (2018) DOI: 10.3390/genes9120571 (Review)
- Simon Chi-Chin Shiu, Andrew B Kinghorn, Yusuke Sakai, Yee-Wai Cheung, Jonathan G Heddle, and Julian A Tanner,* “The Three S's for Aptamer-Mediated Control of DNA Nanostructure Dynamics: Shape, Self-Complementarity, and Spatial Flexibility.” ChemBioChem, 19, 18, 1900–1906 (2018) DOI: 10.1002/cbic.201800308 (Review)
- Satoshi Kimura, Yusuke Sakai, Kenta Ishiguro, Tsutomu Suzuki,* “Biogenesis and iron-dependency of ribosomal RNA hydroxylation.” Nucleic Acids Research, 45, 22, 1–14 (2017) DOI: 10.1093/nar/gkx969
- Akinori Kuzuya,* Yusuke Sakai, Takahiro Yamazaki, Yan Xu, Yusei Yamanaka, Yuichi Ohya and Makoto Komiyama,* “Allosteric Control of Nanomechanical DNA Origami Pinching Devices for Enhanced Target Binding.” Chemical Communications, 53, 59, 8276–8279 (2017) DOI: 10.1039/C7CC03991C
- Yusuke Sakai, Kenjyo Miyauchi, Satoshi Kimura and Tsutomu Suzuki,* “Biogenesis and growth phase-dependent alteration of 5-methoxycarbonylmethoxyuridine in tRNA anticodons.” Nucleic Acids Research, 44, 509-523 (2016) DOI: 10.1093/nar/gkv1470 (NAR Breakthrough Article selected)
- Takahiro Yamazaki, Yuichiro Aiba, Kohei Yasuda, Yusuke Sakai, Yusei Yamanaka, Akinori Kuzuya,* Yuichi Ohya, and Makoto Komiyama*, “Clear-cut observation of PNA invasion using nano-mechanical DNA origami devices.” Chemical Communications, 48, 11361–11363 (2012) DOI: 10.1039/c2cc36358e
- Akinori Kuzuya,* Yusuke Sakai, Takahiro Yamazaki, Yan Xu, and Makoto Komiyama,* “Nanomechanical DNA origami ‘single-molecule beacons’ directly imaged by atomic force microscopy.” Nature Communications, 2, 449 (2011) DOI: 10.1038/ncomms1452
酒井 雄介 講師
Yusuke Sakai
博士(工学)