研究内容の紹介
ビッグデータマイニングによるゲノム動態の機序解明
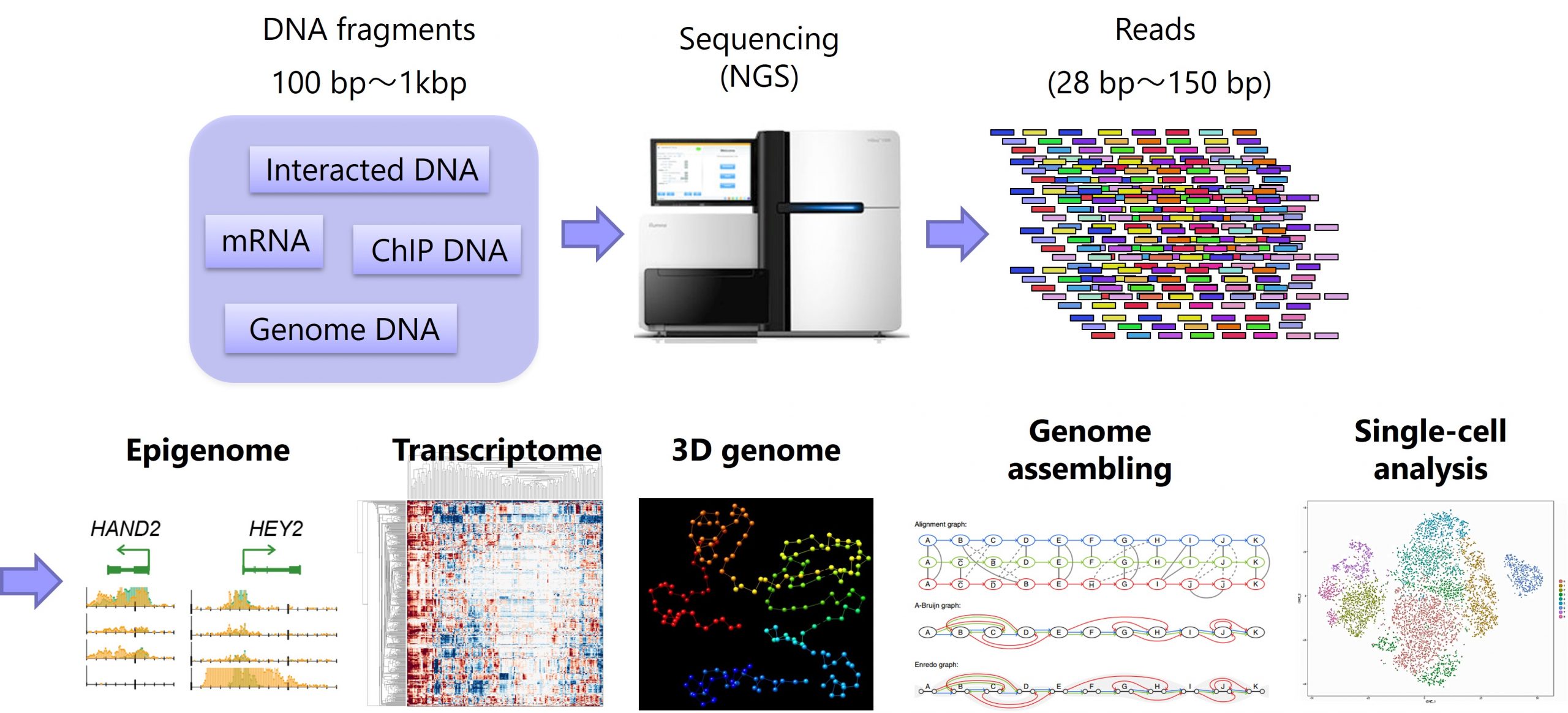
次世代シーケンサ(NGS)を利用したゲノム解析により、転写・複製・立体構造制御などゲノム上のさまざまな動態を直接観測することが可能になりました。国際的プロジェクトにより個人差・がんゲノム・エピゲノムなどの大規模データベースも次々に誕生しており、まさにNGS全盛期と言えます。一方で、生産された膨大かつ多様なNGSデータを統合的に解析し、全く未知の重要な生物学的情報を得る「データ駆動型解析」のために技術開発は未だ発展途上です。
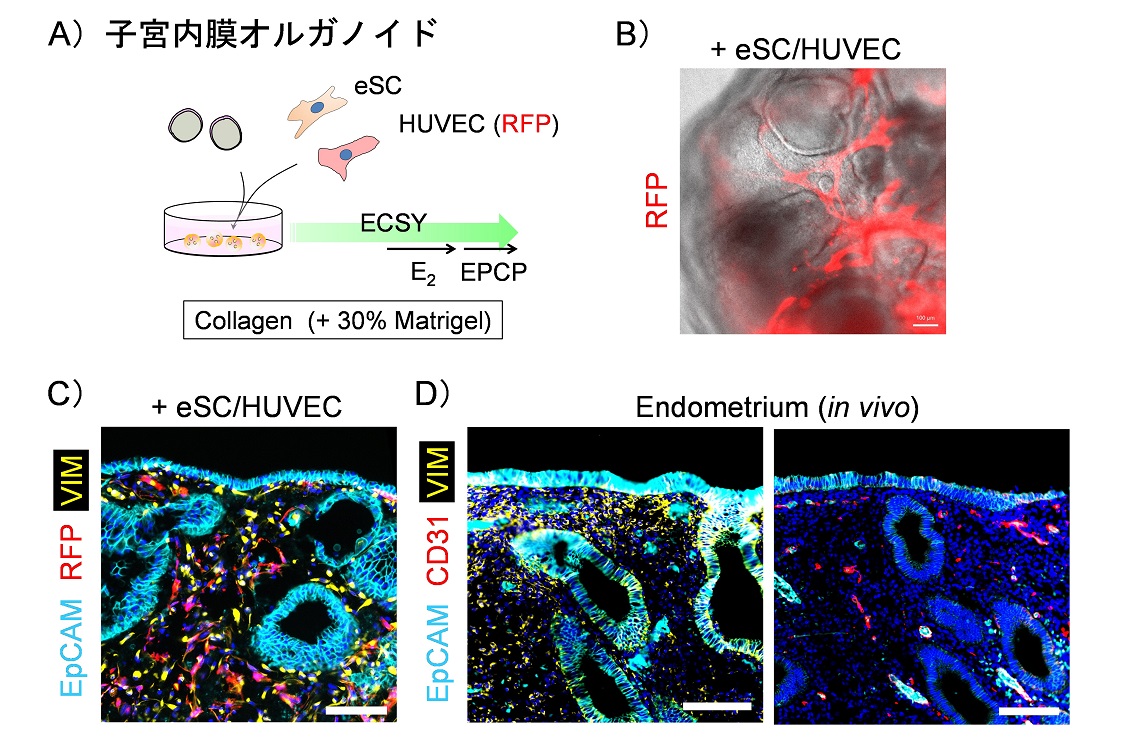
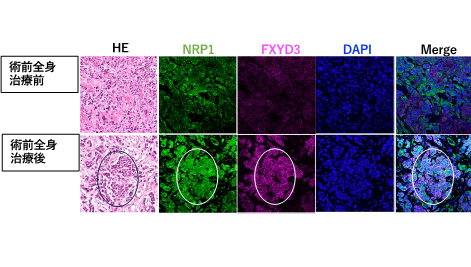
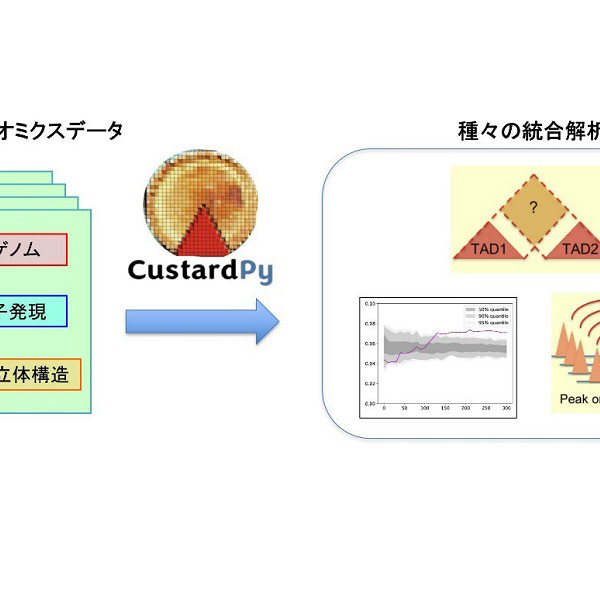
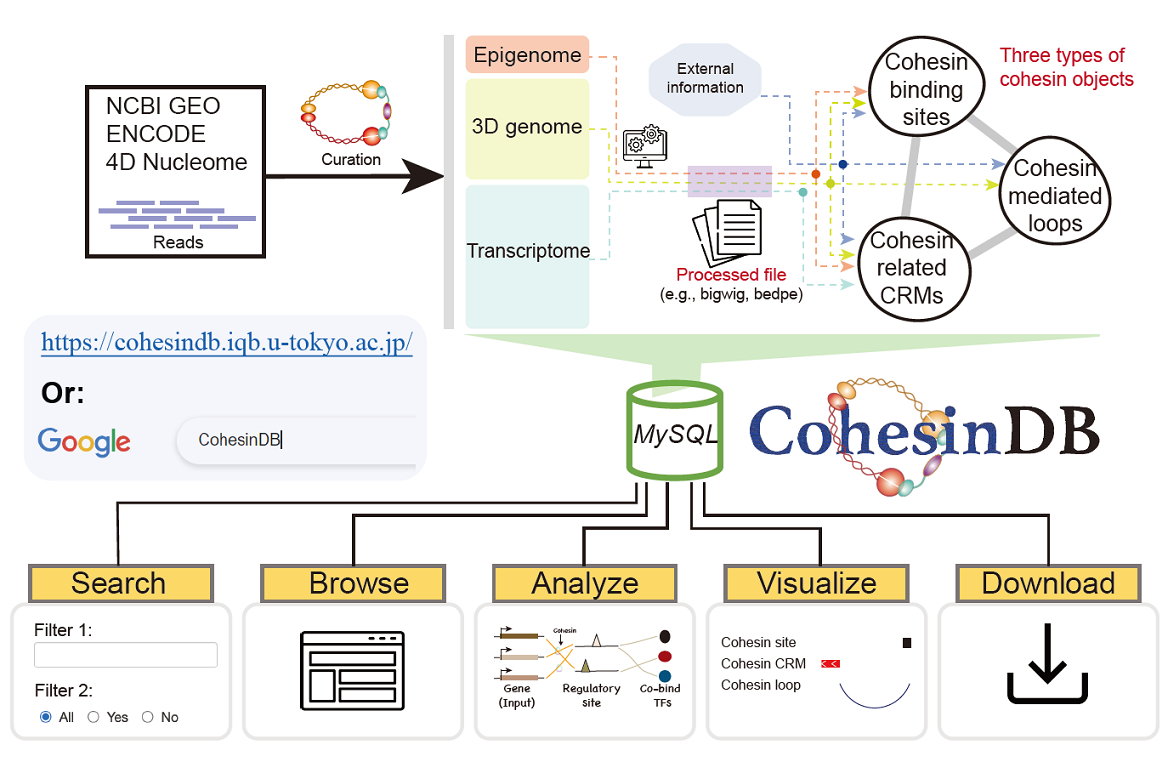
我々はこれまでChIP-seq, RNA-seq, Hi-CなどさまざまなNGSアッセイを利用し、遺伝子疾患患者やノックアウトマウスなどの細胞系を対象に、ゲノム上の制御異常を多面的かつ全ゲノムレベルで研究してきました。ツール開発にも力を入れており、これまでに多サンプルエピゲノム解析のための統合解析パイプラインを構築しています。現在は立体構造、組織内不均一性(heterogeneity)、シミュレーション技術などにも対象を広げ、多様かつ柔軟な解析を可能とする「マルチNGSオミクス・データ駆動型解析システム」の実装に取り組んでいます。