研究内容の紹介
転写とヒストン修飾の生細胞ダイナミクス
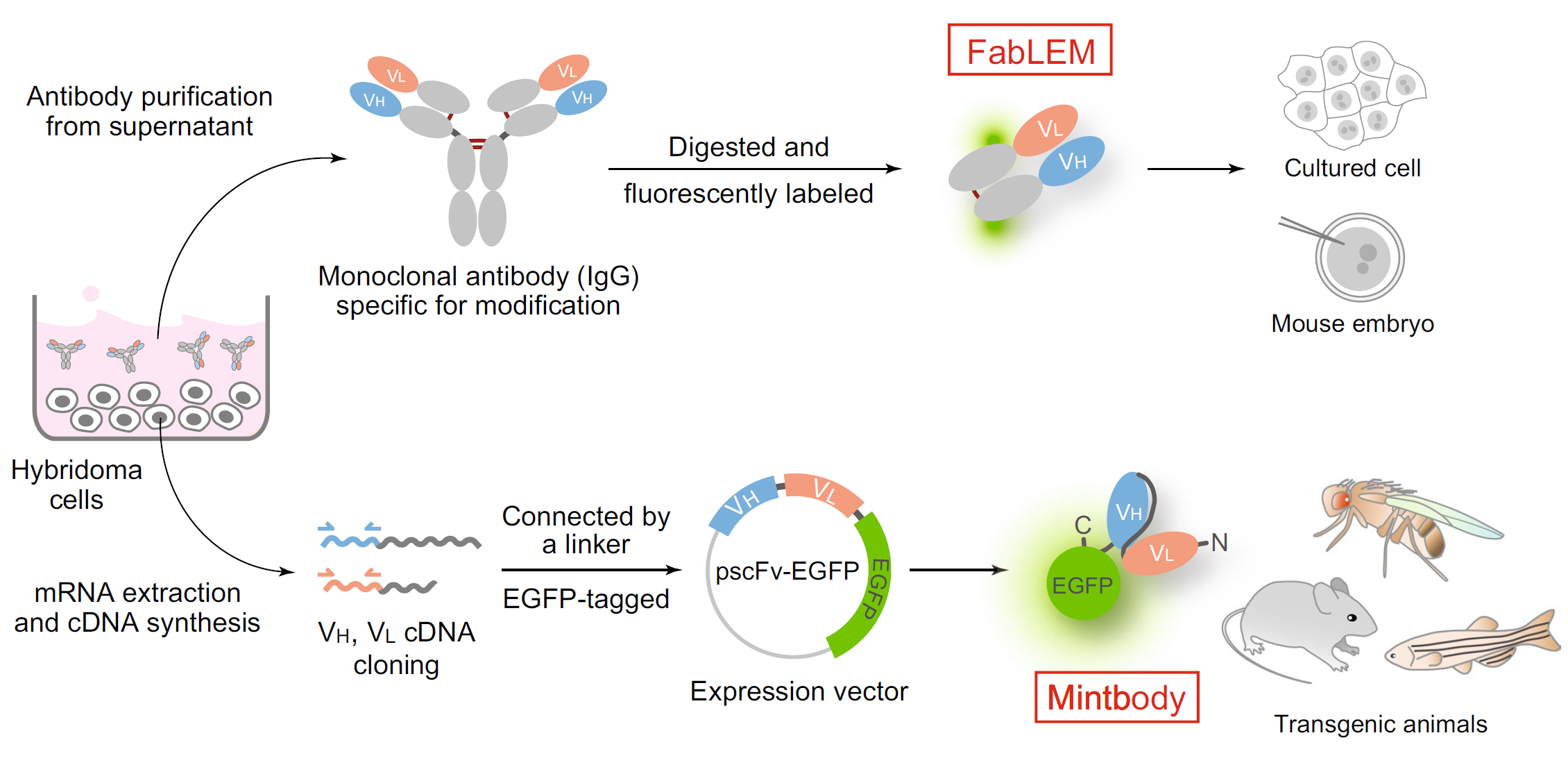
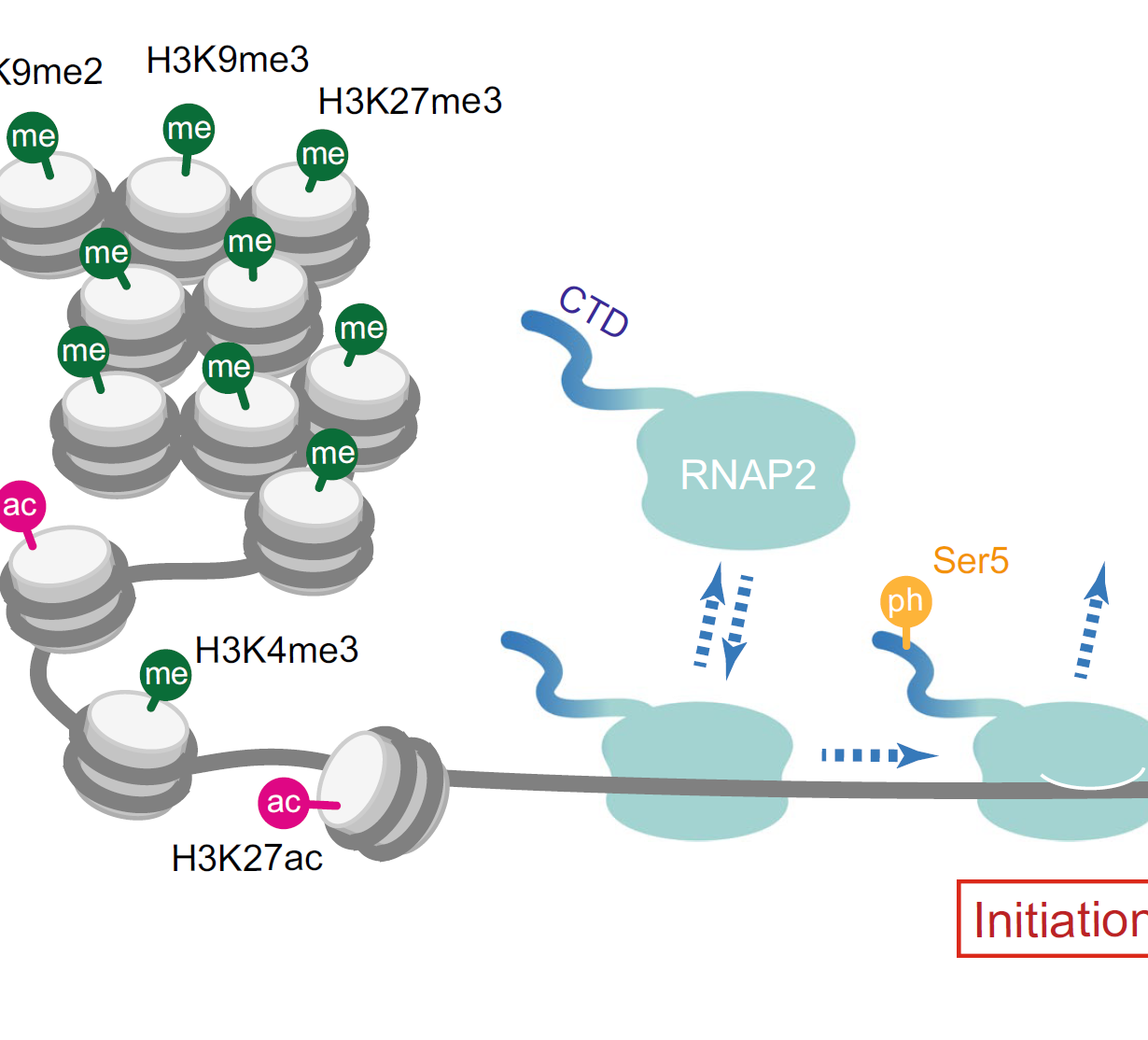
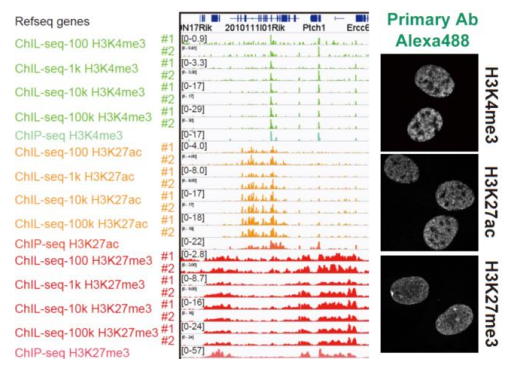
ヒストンやRNAポリメラーゼIIの翻訳後修飾を生きた細胞で検出するため、それらの修飾に特異的なモノクローナル抗体を作製し、抗体由来の蛍光標識抗原結合断片や遺伝子コード型プローブを開発した。特に、遺伝子コード型のプローブは生体内での解析や長時間の解析に有用である。これらの生細胞内修飾可視化プローブを用いて、転写活性化に伴うクロマチン修飾動態の解析を行っている。これまでに、ステロイド系ホルモンによる標的遺伝子アレイの活性化のキネティクスを計測し、ヒストンH3のアセチル化が転写の開始から伸長にいたる過程を促進することを明らかにした。現在、熱ショックストレスによるサテライト遺伝子からの非コードRNAの転写活性化や胚性ゲノム活性化の際のヒストン修飾の変化について計測を行っている。一方、遺伝子発現の抑制に関しては、X染色体の不活性化に伴うクロマチン構造変化を追跡している。また、生細胞解析とエピゲノム解析を融合させるために、イメージングした少数の細胞のエピゲノム情報を取得する方法の開発を進めている。