発表概要
発表のポイント
◆細胞分裂時に、染色体上のDNAループの大きさを適切に制御する仕組みがあること、またこの仕組みが壊れると染色体がスムーズに分離できなくなることを発見しました。
◆DNAループの大きさが、コヒーシンとScc2という2つの因子の相互作用の強さで決定されていることを明らかにしました。
◆本研究の成果は、染色体分配の異常が関与するがんや遺伝性疾患の理解を深め、将来的にはその予防や治療法の開発に貢献することが期待されます。
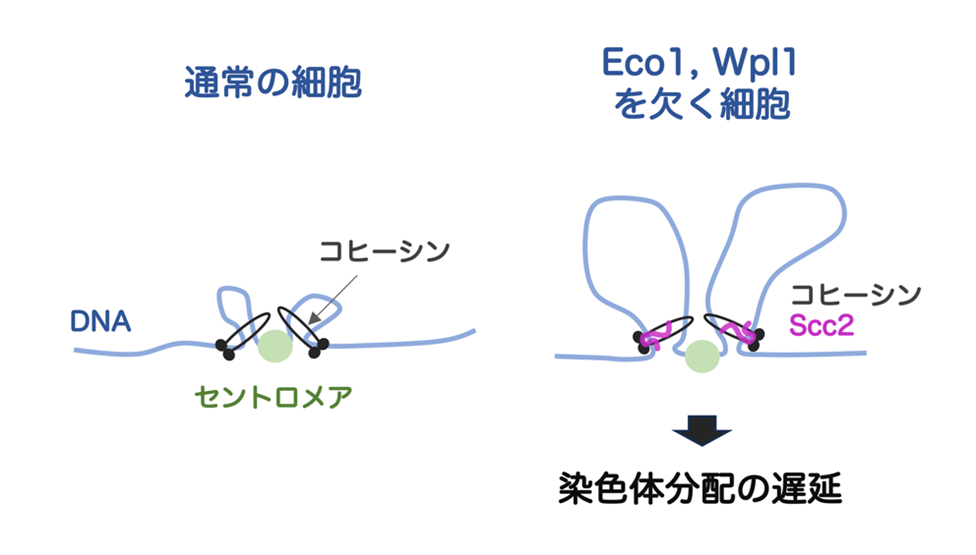
DNAループのサイズ調整が染色体の動きを左右する
発表内容
東京大学大学院総合文化研究科大学院生(研究当時)のSao Anh Nguyen(サオ アィン グェン)さん、同大学定量生命科学研究所ゲノム情報解析研究分野の須谷尚史准教授と白髭克彦教授らによる研究グループは、酵母細胞を使って、細胞分裂時に染色体(DNA)がどのような構造をとっているかについて研究を進めてきました。今回、コヒーシンというタンパク質とScc2(注1)の結びつきが強くなると、染色体に大きな輪(ループ)ができやすくなり、このループが広がりすぎると細胞分裂がスムーズに進まなくなることを明らかにしました。この研究では、染色体の折り畳まれ方を高い解像度で可視化できるMicro-C(注2)という手法を活用しました。
研究の背景と主な発見
【DNAにループを作るコヒーシン】
コヒーシンは、ループを作ることで、長いDNA分子に構造を与えるタンパク質です。哺乳類細胞では、コヒーシンが染色体の折り畳まれ方や遺伝子発現の調整に大きく関わることがわかっています。NIPBLというタンパク質と結合したコヒーシンには、ループの大きさをだんだん大きくする能力があると考えられています。一方、酵母細胞中では、コヒーシンがScc2(ヒトのNIPBLに相当)とほとんど結びついていないため、DNAループはそれほど大きくならないと考えられてきました。
【Eco1とWpl1の協力でループを抑制】
研究チームは、これまでにもコヒーシンの働きを調整する因子として知られていたEco1 とWpl1という2つのタンパク質に注目しました。今回の実験では、Eco1とWpl1を同時に働かなくすると、コヒーシンとScc2の結びつきが強くなり、染色体上でDNAループが大きく広がることを発見しました。すなわち、Eco1とWpl1は、DNAの輪が大きくなりすぎないようにブレーキをかける役割をしていることがわかりました。特に染色体にあるセントロメア(注3)という場所の近傍でループが顕著に拡大しました。
【大きくなりすぎたループは染色体の分配を遅らせる】
研究の結果、大きくなったループをもつ細胞では、細胞分裂過程において染色体が分かれるタイミングが遅れることがわかりました。Eco1とWpl1が両方とも機能しないと、セントロメア近くのDNAループが元の10倍以上の大きさ(数百kb)にまで膨張し、これが分配の進行を阻害する原因となっていました。言い換えれば、Eco1とWpl1が正常に働いている状態では、DNAループが必要以上に大きくならないように保たれ、それによって染色体をスムーズに分配することができるのです。
【Scc2の存在が鍵】
DNAループが大きくなるには、Scc2と結合したコヒーシンの働きによる“ループ押し出し”(注4)という仕組みが必要だと考えられています。今回の実験では、Scc2を人工的に取り除いてしまうと、Eco1とWpl1が存在しない条件でもループが大きく広がらないこともわかりました。つまり、Scc2を呼び寄せる仕組みが変化することで、DNAループの大きさが決定づけられると考えられます(図1)。
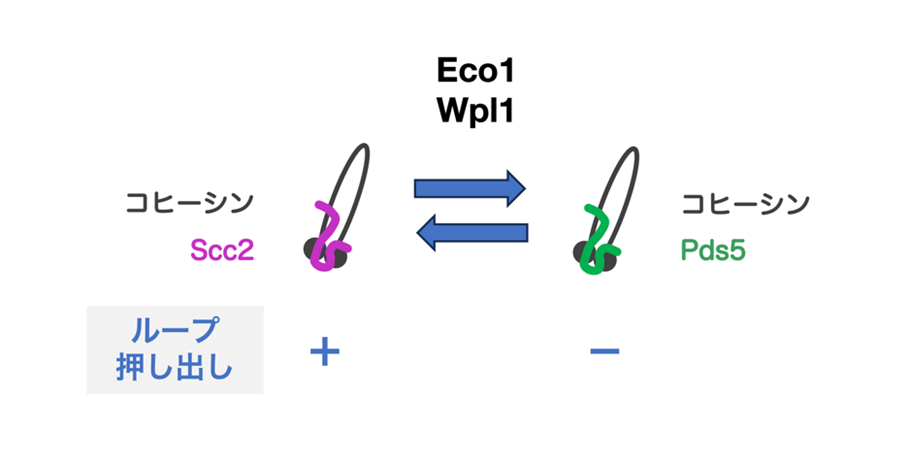
図1:コヒーシンによるDNAループ形成を制御する仕組み
Scc2と結合したコヒーシンは“ループ押し出し”活性を示し、大きなDNAループを形成することができます。Eco1とWpl1が機能するとScc2はコヒーシンに結合できなくなり、ループ押し出し活性が失われます。この時、Scc2に代わってPds5というタンパク質がコヒーシンに結合するようになります。
研究の意味と今後の課題
本研究から、細胞が分裂するときの染色体の形づくりに関わる重要な仕組みが新たに明らかになりました。もしこの制御が崩れると、遺伝情報が子孫細胞にうまく配分されないことが起こりえます。そうした染色体分配の異常は、がんや遺伝性疾患(例えばダウン症候群)の原因の一部になると考えられます。
今回の研究は酵母細胞を用いたものですが、同じ仕組みがヒトの細胞でも働いているかどうかを調べることで、より広く生命の仕組みを理解できると期待されます。
今後は、DNAループを適切なサイズに保つしくみを分子レベルでより詳しく調べることで、がんや遺伝病の原因解明につなげていきたいと考えています。
〇関連情報:
「プレスリリース① コヒーシン複合体による新たな遺伝子発現制御機構の一端を解明―希少疾患や白血病の原因解明の糸口に―」(2022/06/09)
https://www.u-tokyo.ac.jp/focus/ja/press/z0207_00060.html
「プレスリリース② DNA鎖の「よじれ」に対処するメカニズムの発見――Smc5/6複合体は正の超らせんDNAを認識する分子モーターである」(2024/01/31)
https://www.iqb.u-tokyo.ac.jp/pressrelease/20240131/
発表者・研究者等情報
東京大学定量生命科学研究所 先端定量生命科学研究部門 ゲノム情報解析研究分野
須谷 尚史 准教授
白髭 克彦 教授
Sao Anh Nguyen 学術専門職員(サオ アィン グェン)(研究当時:大学院総合文化研究科 広域科学専攻 博士課程)
研究助成
本研究は、日本学術振興会 科研費「JST CREST(課題番号:JPMJCR18S5)」、「学術変革領域研究(A)(課題番号:JP20H05940, JP20H05933)」、「基盤研究(S)(課題番号:JP20H05686)」、「基盤研究(C)(課題番号:JP21K06012)」、および日本医療研究開発機構「AMED BINDS(課題番号:22ama121020j0001)」、「AMED ASPIRE-A(課題番号:JP23jf0126003)」の支援により実施されました。
用語解説
(注1)Scc2:コヒーシンを活性化する作用をもつと考えられているタンパク質。ヒトの同種のタンパク質にはNIPBLという名前がついています。
(注2)Micro-C:細胞の核の中でDNAがどのような構造をとっているか解析するための手法。近接して存在するDNA部位ペアの情報を次世代シークエンサーによって多量に取得することで、高次構造を可視化します。
(注3)セントロメア:細胞分裂のときに染色体の“持ち手”として働く部分です。染色体を正しく子孫細胞に分ける上で極めて重要な役割をもちます。
(注4)ループ押し出し:コヒーシンは柔軟な輪っかのような形態をしたタンパク質複合体です。コヒーシンは、この輪っかの中にDNAの一部をつかみながら「押し出す」ように動くことで、DNAのループをだんだんと大きくしていくと考えられています。この仕組みをループ押し出し(loop extrusion)と呼びます。
アイキャッチ画像
雑誌名等
雑誌名:iScience
題 名:Regulation of pericentromeric DNA loop size via Scc2-cohesin interaction(In Press, Journal Pre-proof)
著者名:Sao Anh Nguyen, Toyonori Sakata, Katsuhiko Shirahige
*, Takashi Sutani
*(*, 責任著者)
DOI: https://doi.org/10.1016/j.isci.2025.112322
URL: https://www.sciencedirect.com/science/article/pii/S2589004225005838?via%3Dihub
問い合わせ先
東京大学定量生命科学研究所 先端定量生命科学研究部門 ゲノム情報解析研究分野
准教授 須谷 尚史(すたに たかし)
Tel:03-5841-7810 E-mail:tsutani@iqb.u-tokyo.ac.jp
東京大学定量生命科学研究所 総務チーム
Tel:03-5841-7813 E-mail:soumu@iqb.u-tokyo.ac.jp





