発表概要
発表のポイント
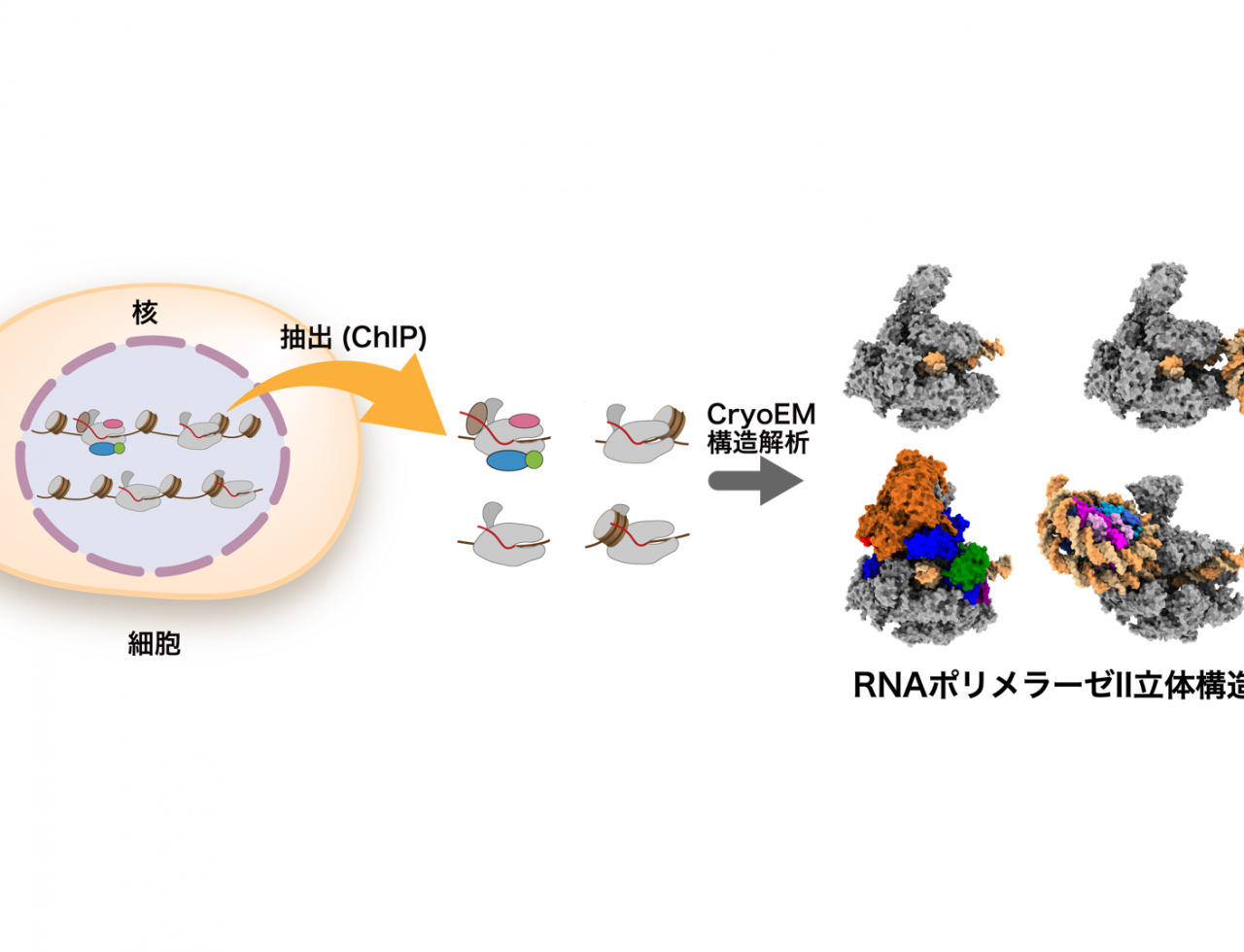
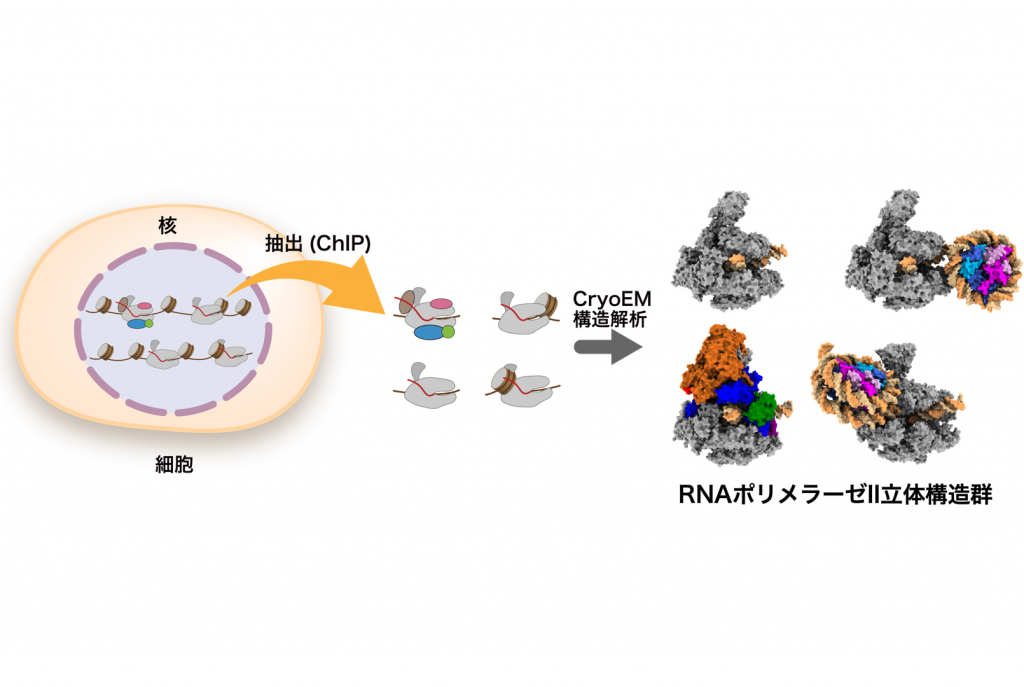
○細胞内のタンパク質をゲノムDNAに結合した状態で抽出し(ChIP)、クライオ電子顕微鏡解析(CryoEM)により可視化するChIP-CryoEM法を確立しました。
○ChIP-CryoEM法により、ヒト細胞内のゲノムDNAをRNAに転写中のRNAポリメラーゼIIの立体構造を可視化することに成功しました。従来の知見を再確認するとともに、新規のRNAポリメラーゼII複合体を発見し、ゲノムDNA転写の新たな機構が明らかになりました。
○ChIP-CryoEM法をさまざまなタンパク質に適用することで、遺伝子の転写のみならず、複製、修復、組換えなどのDNA機能の制御に関する研究が加速し、多様な疾患の原理解明と創薬への貢献が期待されます。

ChIP-CryoEM法による細胞内RNAポリメラーゼIIの立体構造の可視化
発表内容
クロマチン構造機能研究分野の鯨井智也 助教、胡桃坂仁志 教授らによる研究グループは、解析対象のタンパク質をゲノムDNAに結合した状態で抽出する方法(ChIP:Chromatin immunopurification)とクライオ電子顕微鏡解析(CryoEM:Cryo-electron microscopy)を組み合わせた「ChIP-CryoEM法」を確立しました。本方法を用いて、遺伝子発現においてDNAからRNAを合成する酵素RNAポリメラーゼII(注1)の構造解析を行い、ヒト細胞内でゲノムDNAを転写中のRNAポリメラーゼIIの立体構造を可視化することに初めて成功しました。構造解析から、これまで試験管内で再構成されたRNAポリメラーゼIIの立体構造解析の結果を確認したことに加えて、新規のRNAポリメラーゼII複合体構造を発見しました。これらの結果は、RNAポリメラーゼIIの転写機構の解明に対して新たな知見を与えると同時に、細胞内のさまざまなDNA結合型タンパク質の効率的な立体構造解析につながる新たなツールとして寄与することが期待されます。
研究背景
RNAポリメラーゼIIは、遺伝情報が書き込まれたDNAをRNAに転写する中心的な酵素であり、遺伝子の発現の根幹を担っています。そのため、RNAポリメラーゼIIの転写機構の解明は、生命活動の仕組みを理解するうえで非常に重要です。細胞内でゲノムDNAは、ヌクレオソーム(注2)と呼ばれる構造を形成し、ヌクレオソームが連なるクロマチン構造(注3)を形成しています(図1)。細胞内でRNAポリメラーゼIIはクロマチンを形成したDNAを転写していると考えられています。RNAポリメラーゼIIによる転写は、さまざまな因子が結合した複合体を形成することで制御されています。世界中の研究者が、転写機構の解明のためにRNAポリメラーゼIIの立体構造解析を進めており、これまでに非常に多くの知見が得られてきました。しかし、細胞内で実際にどのような複合体を形成して転写するのかについては、いまだ不明な点が多いのが現状です(図1)。
これまでのRNAポリメラーゼIIの転写機構の立体構造研究は、RNAポリメラーゼIIを高純度に精製し、試験管内で人工的にDNA上に結合させる、再構成という方法によって実施されてきました。再構成は、精製した限られた因子を試験管内で組み合わせて複合体を形成させるため、目的のタンパク質の構造と機能を詳細に調べるうえで非常に優れた手法です。しかしながら、再構成のためには、解析対象のタンパク質が細胞内でどのような複合体を形成するのか、という事前情報が必要であり、その取得は非常に困難かつ多くの時間を要します。DNA結合型のタンパク質は他のタンパク質やDNAと複合体を形成してその機能を発揮するため、解析対象の遺伝子の機能を理解するためにも複合体の形成情報は非常に重要です。

図1:RNAポリメラーゼIIはクロマチン構造を形成したDNAを転写する
研究内容
本研究では、細胞内で転写中のRNAポリメラーゼIIを可視化するChIP-CryoEM法を確立し、クロマチンにおける転写機構の理解を深めることを目指しました(図2)。この研究では、細胞内でDNAと結合した複合体の状態でRNAポリメラーゼIIを抽出するため、RNAポリメラーゼIIに精製のためのタンパク質タグ(注4)を付加した細胞を用いました。ゲノムDNAを核酸消化酵素によって分解した後に、精製タグを用いてRNAポリメラーゼIIを精製しました。ゲノムDNAを分解する理由として、RNAポリメラーゼIIは長大なゲノムDNAに結合しており、DNA切断処理なしに抽出を試みると長大なDNAの凝集体として抽出されてしまい、高分解能を実現するクライオ電子顕微鏡の単粒子解析(注5)には適していないためです。その後、精製したRNAポリメラーゼII複合体を、密度勾配遠心分離法(注6)によって凝集体を除去しつつ、化学固定して安定化しました。このように調製した複合体の試料について、クライオ電子顕微鏡の単粒子解析を行いました。

図2:ChIP-CryoEM法の流れ
立体構造解析の結果、ゲノムDNAを転写伸長しているRNAポリメラーゼIIの構造を決定しました(図3a)。また、転写伸長を助ける、転写伸長因子(注7)SPT4、SPT5、SPT6、ELOF1が結合したRNAポリメラーゼII複合体も決定しました(図3b)。この構造は、それぞれの転写伸長因子が一体となって働いていることを示唆しています。DNAを鋳型にRNAが合成された様子が観察できるほど高分解能で解析することができました (図4)。
他にも、RNAポリメラーゼIIとヌクレオソームの複合体の構造2種類を可視化することに成功しました。1つ目は、ヌクレオソームに衝突し、入口で一時的に停止したRNAポリメラーゼIIです(図3c)。この構造によって、これまでにゲノム解析(注8)によって示唆されていた、細胞内ではRNAポリメラーゼIIがヌクレオソームの入口付近で一時停止する、立体構造が実際に確認できました。この一時停止は、これまでの研究からRNAポリメラーゼIIが転写を開始してから最初のヌクレオソームに衝突したときに特に一時停止しやすいと考えられており、遺伝子の転写のON、OFFを制御するという重要な役割を果たすと考えられます。2つ目は転写して通り過ぎたDNA上でヌクレオソームを再構成していると考えられるRNAポリメラーゼIIの構造です(図3d)。この構造は、今までに未報告の構造であり、RNAポリメラーゼIIがヌクレオソームを再構成する機能を持つことを示唆しています。

図3:本研究チームが今回決定したRNAポリメラーゼII立体構造群。
a. RNAポリメラーゼII伸長複合体。b. 転写伸長因子SPT4/5、SPT6、ELOF1結合型RNAポリメラーゼII伸長複合体。c. ヌクレオソームで一時停止中のRNAポリメラーゼII伸長複合体。d. ヌクレオソームを再構成中のRNAポリメラーゼII伸長複合体。

図4:RNAポリメラーゼIIの活性中心におけるDNAとRNAの二重らせんの立体構造
今後の展望
本研究によって、ヒト細胞内で転写中のRNAポリメラーゼIIの立体構造が明らかになりました。これらの立体構造は、RNAポリメラーゼIIによる転写に基づく広範な遺伝子発現制御の理解に重要な知見を与えるものです。そして、今回確立したChIP-CryoEM法は、ゲノムDNAに結合したさまざまなタンパク質複合体について、細胞内に近い状態で立体構造を解析できる方法を提案し、それぞれの構造に基づいた複合体の形成情報の効率的な取得を可能にします。これによって、クロマチンに関連する多様なタンパク質複合体の構造と機能の研究が進み、さまざまな疾患の原因解明に貢献することが期待されます。
付記
本研究は、日本学術振興会(JSPS)の科研費(課題番号:JP20H03201、JP20H05690、JP22K15033、JP23K17392、JP24H00062、JP23H05475、JP24H02328、JP22K06098、JP22KJ0858、JP22J20655、JP22H04696、JP23H04288、JP21H05292、JP23H02394、JP23H00372、 JP24H02323、JP22H04676、JP22K19275、JP20H03182)、科学技術振興機構(JST)の戦略的創造研究推進事業 ERATO(課題番号:JPMJER1901)、同 CREST(課題番号:JPMJCR24T3、JPMJCR24Q1、JPMJCR23N3)、同 さきがけ(課題番号:JPMJPR2026)、日本医療研究開発機構(AMED)の革新的先端研究開発支援事業(AMED-CREST)(課題番号:JP23gm1810008)および生命科学・創薬研究支援基盤事業(BINDS)(課題番号:JP25ama121009、JP23ama121017j0001)の支援により実施されました。
用語解説
(注1)RNAポリメラーゼII
DNAを鋳型にして、RNAを合成することを転写といい、RNAポリメラーゼIIはこの反応を担っている。ヒトのRNAポリメラーゼには3種類あるが、RNAポリメラーゼIIは、遺伝子の発現に関与するRNAの合成を担当する。転写には、RNAポリメラーゼIIがDNAに結合してRNA合成を開始する「開始」フェーズ、RNAを継続的に合成する「伸長」フェーズ、RNAの合成を終了する「終結」フェーズがある。細菌からヒトまで共通する保存された酵素。
(注2)ヌクレオソーム
4種類のヒストンタンパク質H2A、H2B、H3、H4からなる複合体にDNAが巻き付いた構造体。このヌクレオソームがいくつも連なる数珠状構造を骨格としてクロマチン構造が形成される。
(注3)クロマチン構造
酵母からヒトに至るまで、生き物のゲノムDNAは、ヒストンタンパク質に巻き付いて細胞の核内に収納されており、数珠状の構造を取っている。この数珠状の構造には多様なタンパク質が結合しており、その巨大な複合体を「クロマチン」と呼ぶ。クロマチン構造は長大なDNAを効率よく収納するだけでなく、その構造の変換を通して遺伝子の働きを制御している。
(注4)タンパク質タグ
タンパク質精製のために用いられる特定のアミノ酸配列。今回の研究では、FLAGというタグが用いられており、このタグのアミノ酸配列が解析対象のRNAポリメラーゼIIに遺伝子工学的に挿入されている。このタグに特異的に結合する抗体と呼ばれるタンパク質を用いることで、RNAポリメラーゼIIを抽出することができる。
(注5)クライオ電子顕微鏡の単粒子解析
タンパク質などの試料を氷の中に閉じ込め、極低温下で電子線を用いて高解像度の画像を取得する装置である、クライオ電子顕微鏡を用い、タンパク質1分子ずつについて、さまざまな方向を向いた分子の画像を収集し、コンピューターによる画像処理と組み合わせることによって、高解像度の3次元立体構造を決定する。
(注6)密度勾配遠心分離法
試料中に含まれるさまざまなタンパク質を、タンパク質の粒子のサイズ、形状、および密度に基づいて分離する方法。
(注7)転写伸長因子
RNAポリメラーゼIIに結合して、転写伸長反応を助ける因子。RNAポリメラーゼIIの転写の開始、伸長、終結のフェーズのうち、伸長のステップで機能する。
(注8)ゲノム解析
生物のゲノム配列を網羅的に解析し、それぞれの配列にどのような特徴があるのかを調べる手法。注目している因子のゲノム配列中での結合場所や、遺伝子の発現量などを明らかにすることができる。
アイキャッチ画像
雑誌名等
雑誌名:Nature Communications
題 名:Multiple structures of RNA polymerase II isolated from human nuclei by ChIP-CryoEM analysis
著者名:Tomoya Kujirai(鯨井智也), Junko Kato(加藤淳子), Kyoka Yamamoto(山本京香), Seiya Hirai(平井誠也), Akihito Harada(原田哲仁), Lumi Negishi(根岸瑠美), Mitsuo Ogasawara(小笠原光雄), Yuki Yamaguchi(山口雄輝), Yasuyuki Ohkawa(大川恭行), Yoshimasa Takizawa(滝沢由政), Hitoshi Kurumizaka(胡桃坂仁志)*
【*:責任著者】
DOI:10.1038/s41467-025-59580-x
問い合わせ先
(研究内容については発表者にお問合せください)
東京大学定量生命科学研究所
教授 胡桃坂 仁志(くるみざか ひとし)
Tel:03-5841-1467 E-mail:kurumizaka@iqb.u-tokyo.ac.jp
東京大学定量生命科学研究所 総務チーム
Tel:03-5841-7813 E-mail:soumu@iqb.u-tokyo.ac.jp
科学技術振興機構 広報課
Tel:03-5214-8404 E-mail:jstkoho@jst.go.jp
(JST事業に関するお問合せ)
科学技術振興機構 研究プロジェクト推進部 ICT/ライフイノベーショングループ
今林 文枝(いまばやし ふみえ)
Tel:03-3512-3528 E-mail:eratowww@jst.go.jp