「命のプラットフォームとしての染色体の構造と機能をゲノム学を通して解き明かす」
ということになります。 生物の生存・増殖のためには染色体(=遺伝情報)が子孫へ間違いなく受け継がれてゆくことが不可欠です。生命はこの目的のために染色体を複製し、生じた損傷を修復、倍化した染色体を子孫細胞へと分配する分子機構を生み出してきました。染色体は遺伝情報の発現の場であるとともに、これら遺伝情報維持に関与する数百、数千のタンパク質因子が活躍する場でもあります(図1)。
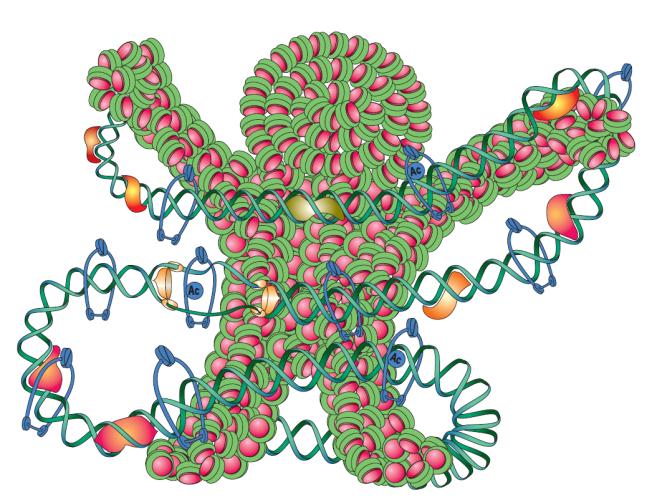
図1 生命のプラットフォームである染色体の概念図
あるタンパク質因子が染色体のどこで機能しているのかを塩基レベルの解像度で決定する手法としてChIP-chip( chromatin immunoprecipitation on DNA chip) 法があります。我々は世界にさきがけてタイリングアレイ(ゲノムの隅から隅まで余すところなく解析可能な、集積度、プローブ密度の高いDNA chip)を導入し、高精度にタンパクの染色体上での動態を解析可能な技術を構築しました。そして、この手法を用いて染色体維持に関わる因子が染色体のどこで機能しているか、その局在は細胞周期や様々な変異体中でどう変化するか、という動態を明らかにしてきました(図2、図3)。染色体上で機能している現場を直に可視化することで、それぞれの因子が果たしている役割の本質を知ることができます。また、様々な因子の結合プロファイルを比較することで、これまで予想されていなかった因子間の機能連関を明らかにすることにも成功してきました(業績欄参照)。
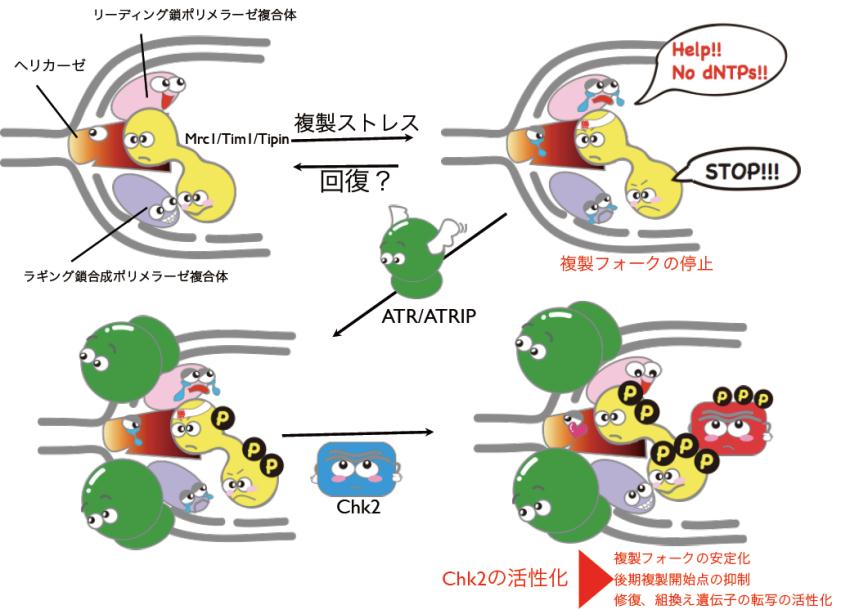
図2 過去のChIP-seq、ChIP-chip解析から明らかとなったチェックポイント因子による複製フォークの制御
(Katou et al. nature, 2003および、De Piccoli et al, Mol Cell, 2012に基づく)

図3 コヒーシンとCTCFによる転写インシュレータの構築。写真はnatureのNews & Viewsで紹介されたもの。
ゲノム解析技術は「次世代DNAシークエンサー」の登場により今大きく様変わりしています。この大量並列型シークエンサーとChIP法を組み合わせる(ChIP-seq法)ことで、ヒトのような巨大で複雑な染色体でも正確に感度よくタンパク質局在を決定できることが分かってきました。特に今までDNA chipでは解析が困難であった繰り返し配列について知見を得ることが可能になったことは、染色体研究に取って大きな進歩であると言えます。私達の研究室では、今後このChIP-seq法の実験・解析技術をさらに応用し、これまで解析の難しかった因子やヒトに特有の因子の機能解析をすすめていこうとしています。また、核内でのDNAの空間的配置を調べる新手法(Hi-C、ChIA-PET等)とChIP-seq法の結果を組み合わせて、「染色体はどのように折り畳まれているのか」という細胞生物学の長年の疑問にも迫りたいと考えています。
私たちの研究室の20年来のテーマは、真核生物の一本の染色体が複製される際に染色体上で生じる全てのことを詳細に記述したいということでした。この研究は1993年までさかのぼりますが、この目的の為に、出芽酵母の2番目に短い第六染色体から全ての複製開始点の候補を系統的に単離し、個々の開始点の特異性を解明するという仕事を始めました。当時はゲノム配列も未だ明らかになっていなかった時代ですので、まさに酵母染色体の物理地図の上に複製開始点の候補断片を配置するという仕事が出発点でした。その後、モデル生物のゲノム配列が解読され、アレイや次世代シークエンサー等の網羅的解析技術が登場し、今では一本の染色体に限らず全ての染色体の端から端まで、その気になれば数時間で、タンパクの配置、DNA複製領域について高解像度の解析結果を得ることが可能になりました。この染色体複製の制御の解明と言うテーマは未だに当研究室の中心的なテーマです。今までに、複製チェックポイント蛋白質と呼ばれる一群のタンパクがどのように染色体複製の特に伸長反応を制御しているのかに焦点を当てた研究を行ってきましたが、最近では特に複製中の染色体上にどのようにして姉妹染色分体間接着因子(コヒーシン)がロードされ、どのようにして姉妹染色分体間接着は確立するのかを解明すべく解析を行っています(図4)。
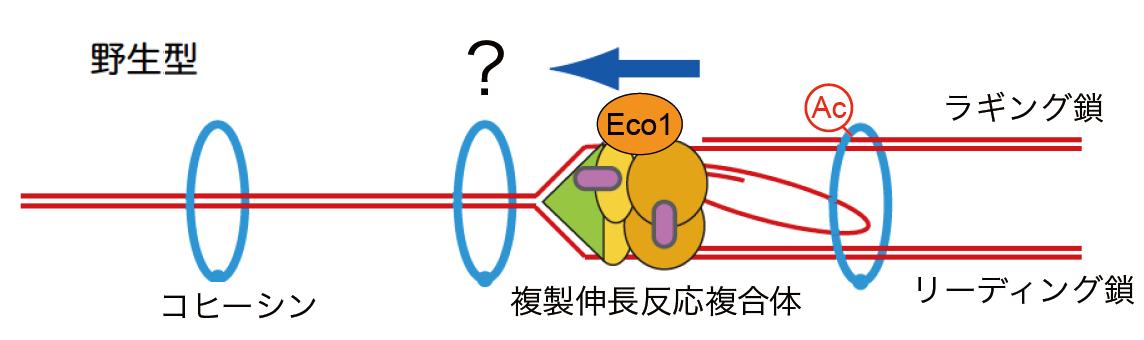
図4 謎が多い、複製フォークによる姉妹染色分体間接着の確立過程。
姉妹染色分体間接着確立には図に示したようにコヒーシンのアセチル化が重要な役割を果たす。
第二のテーマはSMC (Structural Maintenance of Chromosomes)タンパクの機能を探ることです。SMC タンパク質は、6つのファミリー(SMC1からSMC6)に分類されます。先に述べた、コヒーシンタンパクにはSMC1とSMC3が含まれ、姉妹染色分体の接着を通し、染色体分配に必須の機能を有しています。SMC2とSMC4は染色体凝縮に関わるコンデンシンとして機能します。 SMC5とSMC6は染色体のトポロジーを制御していると考えられていますが、その詳細は明らかではありません。私たちはそれぞれのファミリーの染色体上での配置を明らかにすることで、その機能の手がかりを得ようとしてきました。その結果、コヒーシンについては面白いことに、その分配機能とは独立に転写のインシュレーターとして転写制御にも機能していることを発見しました。以前からコヒーシン自身、あるいはコヒーシンを染色体にロードする為に必要なNIPBタンパクの変異はハエでは羽の形成異常が引き起こされること、また、ヒトの遺伝病であるコルネリア•デ•ランゲ症候群(重篤な発生、分化異常を特徴とする疾患)の6割の原因はNIPBL(NIPB like)タンパクであることが判明しており、コヒーシンが高等真核生物では転写にも関与するのではないかと考えられていました。私たちは、この疾患患者の細胞では染色体上のコヒーシンタンパクの量が減少していることが共通の特徴であることも示し、コヒーシンによる転写制御機構の分子機構の一端を明らかにしました。現在、私たちは、コヒーシンローダー、コヒーシンのアセチル化修飾と転写制御の関係、また、コンデンシンタンパクと転写の関係についてゲノム学的手法を用いて解析を進めています。また、SMC5と6タンパクについても、その染色体上での配置から機能の手がかりを解明しようとしています。
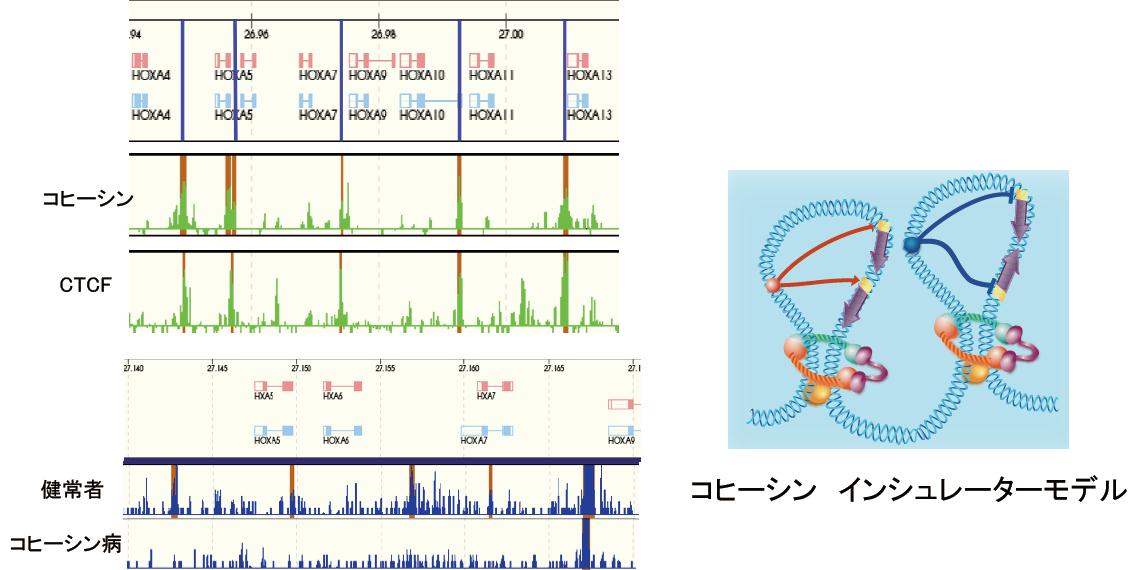
図5 ChIP-seqによる、コヒーシンとCTCFの共局在(左上)とコヒーシン病患者におけるコヒーシン結合サイトの喪失(左下)、コヒーシンループモデル(右)
(Wendt et al. nature 2008, Liu et al., PLoS Biol. 2009, Deardroff et al., nature 2012 による)
第三のテーマは染色体情報学とでも言うべきものです。現在、私たちは出芽酵母、分裂酵母、マウス、ヒトを中心にそれぞれ染色体上に300以上のDNA結合タンパク質の結合プロファイルを分析しています。 これらデータの比較解析から、大きく複雑な真核生物ゲノムの維持のための染色体構築、制御の違いを情報学的に明らかに出来ると考えています。最終的には、それぞれのモデル生物について、より高次の染色体構造、動態の違い、機能的連携について明らかにし、モデル化することが目的です。実践的には次世代シークエンサーから得られたデータをより簡便に、より直感的に扱えるプログラムの開発を手がけています。ここで言う所の簡便、直感的というのはとにもかくにも最短で解析結果を可視化する為のプログラムという意味です。もちろんデータをより深く解析することは本来のテーマですが、ここでは、実験研究者が次の実験の指針を早く得る為の、誰でも中身を知り、取り扱える汎用性の高いプログラムの開発を行っています。
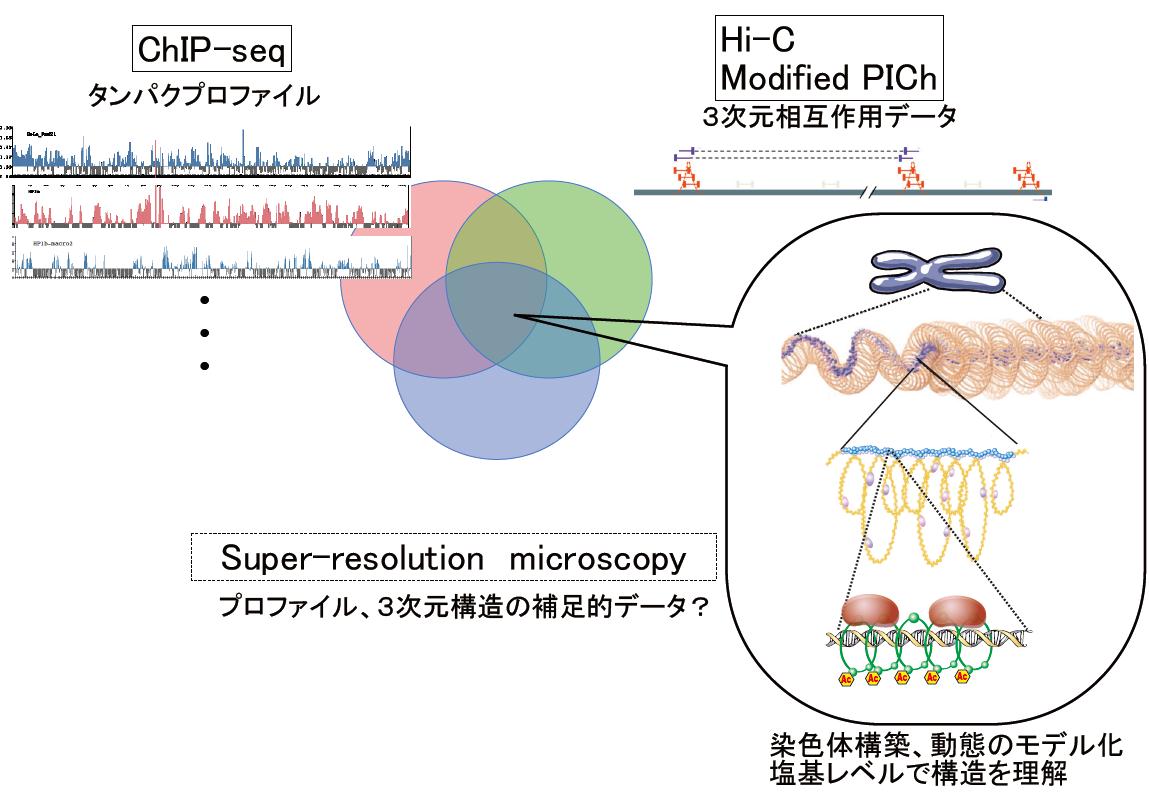
図6 染色体情報学に基づく染色体動態のモデル化


