ラボマニュアル
Manuals
PyMOLの入手方法
PyMOLはver.1以上の新しいものも出ていますが、登録なしで使うにはver.0.99など、古いバージョンをダウンロードします。
http://www.pymol.org/ (外部リンク)
の上にある ”Download”をクリック。
次に、“.... Older PyMOL builds (pre-1.0) are still Freely downloadable...” の部分のリンクからダウンロードページに進んでください。
ちなみに、”evaluation only”は、図の出力の際に画像に”evaluation only”と文字が入ります。
PyMOLの使い方
PDBファイルを読み込んで、リガンドの周囲を観察する場合を想定して使い方をまとめました。
PyMOLリガンド周囲の観察とラベル.pdf (1.4MB)
タンパク質同士の重ね合わせ(align)や、見た目の設定の仕方、図の書き出し方等を説明してあります。そのうち分かりやすいように書き直します。
ファイル形式
PyMOLを使う上で、PDBファイル「.pdb」とpseファイル「.pse」の違いはしっかりと把握しておきましょう。PDBファイルは、座標を含むもので、PyMOL以外のソフトでも自由に開けます。一方、pseファイルはPyMOL sessionファイルと呼ばれるもので、PyMOLでしか開けません。また、pseファイルは座標ファイルではなく、「命令(コマンド)の集合」のようなものです。どのPDBファイルを読み込んで、どの部分の色を変えるか、どのような角度で表示するか、などの命令(操作)が記述されたファイルと考えてよいと思います。
二つのタンパク質、たとえばPPARαとPPARδの重ね合わせをしたいとします。重ね合わせをするには、それぞれのPDBファイルを読み込んでalignします(alignの具体的な方法は前述のpdf参照)。PDBファイルではなく、PPARαとPPARδそれぞれを表示させたpseファイル同時に読み込んで重ね合わせる、と言った事はできません。PPARαのpdbファイルを読み込んだ状態で、例えばPPARδのpseファイルを開いたら、PPARδが(pseに保存されているように)表示されるだけです。
ファイルの保存 (.pse, .pdb)
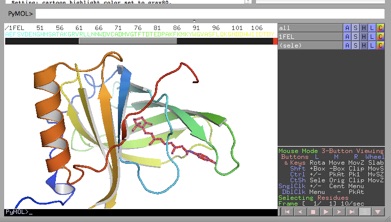
pseファイルとして保存するには、メニューバーのFile > Save Session Asで保存できます。PDBファイルとして出力する場合はFile > Save Moleculeから保存します。ただし、PDBファイルに出力できるのは、一つの”object”(下の図で言うと “1FEL” )、あるいは、一つの”selection”(図の ”(sele)” )です。複数のobjectをまとめて一つのPDBファイルにする場合は、あらかじめ複数のobjectを一つのobjectにまとめておくか、一つのselectionにまとめておく必要があります。
図の書き出し
図を書き出すには、File > Save Imageとします(デフォルトではPNGファイルとして保存されます)。基本的には、図を書き出す前に、”ray” (マウスでクリック or コマンドラインで ”ray” )をしてきれいに描画させてから保存します。図のサイズを指定したい場合は、”ray 1000, 1200”とすれば、1000 pixels × 1200 pixelsのサイズで描画されます。
PDBファイルの編集
たまに、PDBファイル中にリガンドが二つ重なっている場合があります。下のファイルは、そのような場合に片方のコンフォメーションのリガンドのみ表示する方法を説明しています。また、PDBファイルの”中身”(形式)についても触れていますので、参考にしてください。
PyMOLリガンドがダブる場合の処理.pdf (200 KB)
PyMOL (PDB viewer) マニュアル
10/07/23
